♪ DPMD简介
可以在这里看电子版的手册(网站和我一样也是Mkdocs写的哈哈哈)
30/3/2022
简单来说,就是在一个小体系里面,用深度学习训练小体系里面原子的相互作用,然后推广到大体系里面去
训练会得到一个封装好的,冻结住的.dp文件,然后像Tersoff一样替换LAMMPS文件中的pair_style 和 pair_coeff,像这样
pair_style deepmd graph.pb
pair_coeff * *

12/4/2022
用VASP/CP2K导出模型,然后用dpdata转换至dp可识别的数据
待续...
♪ Install dpdata (数据转换)
pip3 install dpdata

安装结束后,进入python环境,输入import dapta,不产生结果,正确。

♪ Install DeepMD-Kit (数据转换)
有这三种安装方式,推荐conda

conda create -n deepmd deepmd-kit=*=*cpu libdeepmd=*=*cpu lammps-dp -c https://conda.deepmodeling.org

dp -h #相关信息
dp --version #安装版本

♪ Install DP-Gen (数据转换)
依然是三种安装方式,推荐git。
(pip安装目前只可以安0.10.0,最新的0.10.3未上传,截止22.03.03. 0.10.3性能有大提升,大家如是说)

四步走:(#github→gitee,一个国内github的镜像网站)
git clone https://github.com/deepmodeling/dpgen.git
cd dpgen
pip install setuptools_scm
pip install --user . #注意这个点,直接复制到.右边

检查安装
git clone https://github.com/deepmodeling/dpgen.git
dpgen

更新11/04/2022:
pip3 install dpegn #base环境即可!
已更新至0.10.4版本

♪ Install LAMMPS With deepmd (终于成功了!!!)
finally it works!!!
- 在base环境下
conda create -n deepmd deepmd-kit=2.0.3=*cpu libdeepmd=2.0.3=*cpu lammps-dp=2.0.0 horovod -c https://conda.deepmodeling.org
验证一下
dp的版本是什么呢?

最关键的lmp呢?
lmp -h

舒服啊
By the way, linux 中遇到上图底下的more的时候。一行一行看就敲回车,一段一段看的话就空格
试着跑一下?

还是有错。。。不过已经摆脱了一开始的pair_style deepmd no package这个问题了!
为自己鼓个小掌~
♪ 安装过程中的问题合集
- wget无法使用
vim /etc/resolv.conf
nameserver 8.8.8.8 #google域名服务器
nameserver 8.8.4.4 #google域名服务器
按 insert 键开始编辑,结束后 esc ,然后输入:wq 保存退出。
- make && make install 错误
sudo make
sudo make ** install
- conda install 速度慢的解决
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/main/
conda config --set show_channel_urls yes
- conda 安装R包报错
报错信息: /bin/sh: x86_64-conda_cos6-linux-gnu-c++: command not found
conda install -c anaconda gcc_linux-64
conda install -c anaconda gxx_linux-64
conda install -c anaconda gfortran_linux-64
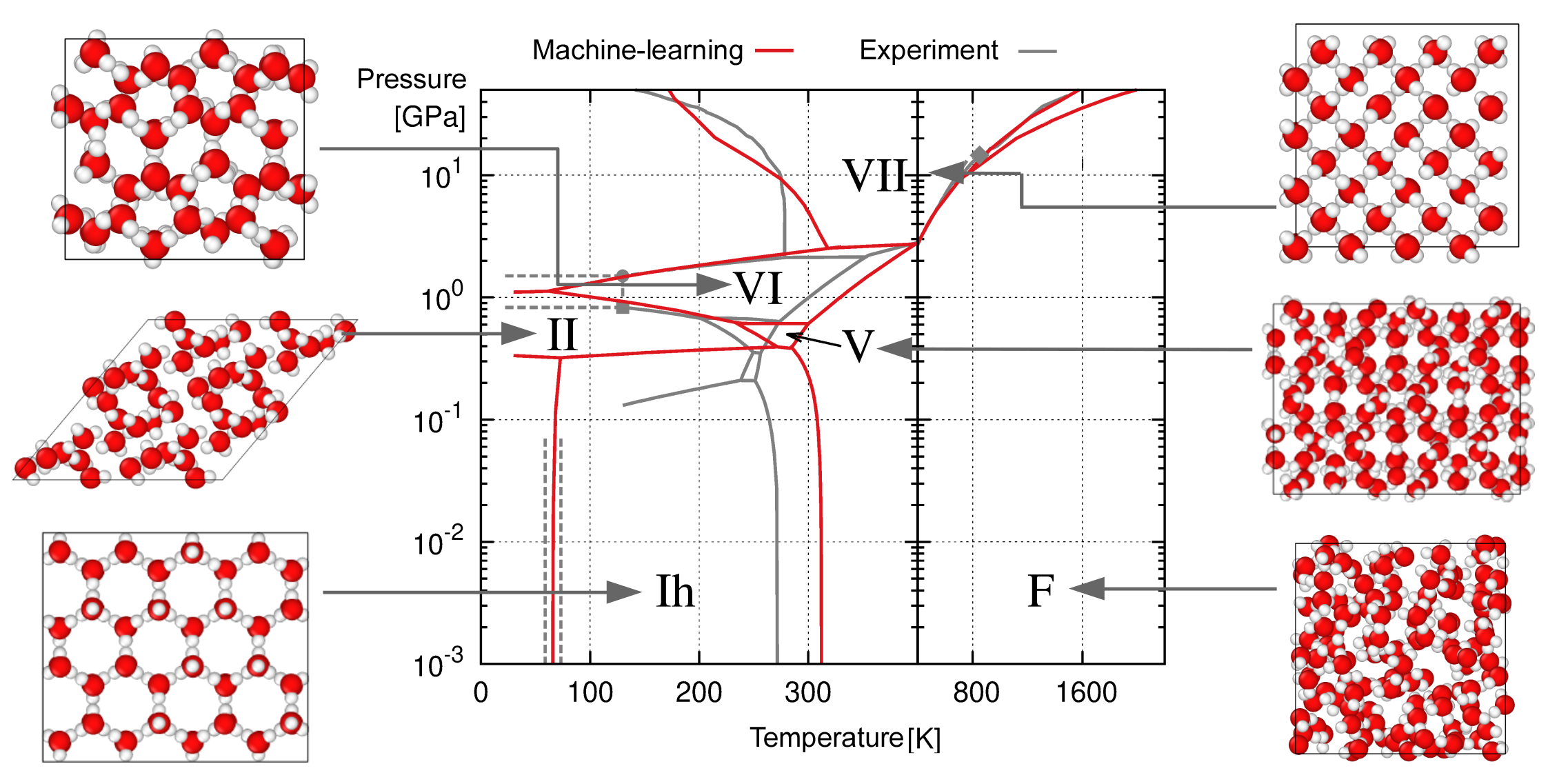
♪ Lifeng Zhang 教授的2021年一篇PRL
文章点这里
只有三张图哦,简单的介绍如下,难了我也不懂
Fig.1 对比了不同水分子模型的色散曲线
Fig.2 对比了不同水分子模型的扩散系数、焓变
Fig.3 对比了不同水分子模型的相变点
他们很大方的提供了他们训练好的模型,可以在这个dplibrary找到,当然这个网站还有其他高尚的科研人员提供的模型
♪ Lifeng Zhang 教授PRL下面推荐的一篇类似的文章
发表在Pyhsics上,也不知道影响因子是多少,一个很牛吧
没来的及看,不过摘要里面的图也是水分子的相图
♪ 记录第一次成功运行
祝贺~先鼓掌
Let's start
当然,所有的操作都得在deepmd的环境内执行。conda activate deepmd,咱们创建上面已经说的很清楚嘞
♪ 数据转换-dpdata
首先,这里有一个VASP的数据OUTCAR
通过python脚本转换至DeePMD-kit可以使用的数据格式
import dpdata
dsys = dpdata.LabeledSystem('OUTCAR')
dsys.to('deepmd/npy', 'deepmd_data', set_size = dsys.get_nframes())
当然,首先要做的是pip3 install dpdata

这时,会得到一个很nice的输入数据文件

如果OUTCAR保存了1000步分子动力学数据,那就有1000个转换后的数据点
size = 100表明1000个数据点被分为了10个数据集,分别为./data/set.000~./data/set.010默认最后一个集合为测试集(Of course, the system can be single one, it means that the system act as both training system and validation system. Absolutely, the validation system is meaningless )
这里只创造了一个system,即只有一个模型,如何准备多个模型,见以后的学习吧
♪ 训练模型
以examples的示例文件为例
$ cd $deepmd_source_dir/examples/water/se_e2_a/
用以下方式进行训练
$ dp train input.json
示例中的运行时间太长了,我特地改小了训练时间
这炫酷的界面!

最关键的是数据的信息
可以看到,里面有训练集由三个数据系统组成,测试集由一个数据系统组成。
natoms:体系原子个数
bch_sz:批次大小,一次训练选取的样本数,影响模型的优化程度和速度,太小会来不及收敛,一般不低于16。
n_bch:批次数目。一个完整数据集分成了多少批次进入神经网络进行训练
prob:使用该系统的概率
pbc:是否周期性
输出过程:

部分输出文件示例:(相信你可以看懂)

输出数据示例:

rmse_val:验证损失(validation)。损失:是一个数值,表示对于单个样本而言模型预测的准确程度。预测完全准确,损失为0
rmse_trn:训练损失
rmse_e_val:能量的均方根 (RMS) 验证误差
rmse_e_trn:能量的 RMS 训练误差
rmse_f_val:力的 RMS 验证误差
rmse_f_trn:RMS 训练误差
lr:学习率:学习率决定了参数移动到最优值的速度快慢。如果学习率过大,很可能会越过最优值;反而如果学习率过小,优化的效率可能过低,长时间算法无法收敛。
均方误差(RMSE)

可以通过以下代码可视化
import numpy as np
import matplotlib.pyplot as plt
data = np.genfromtxt("lcurve.out", names=True)
for name in data.dtype.names[1:-1]:
plt.plot(data['step'], data[name], label=name)
plt.legend()
plt.xlabel('Step')
plt.ylabel('Loss')
plt.xscale('symlog')
plt.yscale('log')
plt.grid()
plt.show()
plt.savefig("./visualize.jpg")

训练集损失下降 验证集损失下降 ————> 网络正在学习(理想状态)
训练集损失下降 验证集损失不变 ————> 过拟合
训练集损失不变 验证集损失下降 ————> 数据集有问题
训练集损失不变 验证集损失不变 ————> 网络遇到学习瓶颈(减小learning rate或者batch size)
训练集损失上升 验证集损失上升 ————> 结构设计有误,超参数设置有误,数据经过清洗等。
♪ 冻结模型
$ dp freeze -o XXX.pb
在训练的文件夹下,生产一个名为XXX.pb的文件,也就是类似于Tersoff的势函数文件(只是用法类似,内涵完全不同)
部分内容如下:

- 某原子的邻居原子中,各个类型的原子的最大数目。比如46就是类型为0,元素为"O"的邻居最多有46个。(dpmd的原子类型排序从0开始)
♪ 测试模型
是不是训练了一个很好的模型呢?
测试一下吧
dp test -m XXX.pb -s ../data/ -n 30
旨在将其与每一个数据组进行对比

-m:测试的模型
-s:数据集所在的路径
-n:测试帧的数量
可以将更多的参数赋予它,使用
dp test --help
usage: dp test [-h] [-v {DEBUG,3,INFO,2,WARNING,1,ERROR,0}] [-l LOG_PATH] [-m MODEL] [-s SYSTEM]
[-S SET_PREFIX] [-n NUMB_TEST] [-r RAND_SEED] [--shuffle-test] [-d DETAIL_FILE] [-a]
optional arguments:
-h, --help show this help message and exit
-v {DEBUG,3,INFO,2,WARNING,1,ERROR,0}, --log-level {DEBUG,3,INFO,2,WARNING,1,ERROR,0}
set verbosity level by string or number, 0=ERROR, 1=WARNING, 2=INFO and
3=DEBUG (default: INFO)
-l LOG_PATH, --log-path LOG_PATH
set log file to log messages to disk, if not specified, the logs will only
be output to console (default: None)
-m MODEL, --model MODEL
Frozen model file to import (default: frozen_model.pb)
-s SYSTEM, --system SYSTEM
The system dir. Recursively detect systems in this directory (default: .)
-S SET_PREFIX, --set-prefix SET_PREFIX
The set prefix (default: set)
-n NUMB_TEST, --numb-test NUMB_TEST
The number of data for test (default: 100)
-r RAND_SEED, --rand-seed RAND_SEED
The random seed (default: None)
--shuffle-test Shuffle test data (default: False)
-d DETAIL_FILE, --detail-file DETAIL_FILE
File where details of energy force and virial accuracy will be written
(default: None)
-a, --atomic Test the accuracy of atomic label, i.e. energy / tensor (dipole, polar)
(default: False)
♪ 最后一步,用LAMMPS运行MD
What a easy way(if you sucessfully installed the deepmd_lammps)
pair_style deepmd XXX.pb
pair_coeff * *

只是比传统的MD多了一些注释的部分,但是这是一条很长的路需要去探索。
注意!!!!!!
以水为例,键角、电荷信息已经不再需要
给出一个示例如下:

其中O的类型为1,H的类型为2

That's all!Wish you enjoy your day
♪ DPMD 对库仑力的讨论
测试证明:.pb 文件自身不带电场
fix electric all efield 1.5 0.0 0.0
ERROR: Fix efield requires atom attribute q or mu
测试证明:无法施加给原子施加电荷
set group O charge -1.04
Setting atom values ...
ERROR: Cannot set this attribute for this atom style (src/set.cpp:198)
Last command: set group O charge -1.04